Introducción
El objetivo de este post es aplicar todo lo posible sobre el 🚢 dataset de Titanic. Al ser un dataset muy fácil de interpretar, se busca que los conceptos sean entendidos fácilmente.
➡️Para más información sobre este dataset: Competencia de Kaggle: Titanic - Machine Learning from Disaster
Show code
xaringanExtra::use_panelset()1. Librerías y definiciones
Se cargan las librerías a utilizar.
Show code
library(tidyverse)
library(gt)
library(gtsummary)
library(skimr)
library(tidymodels)
library(modelsummary)
library(equatiomatic)
library(marginaleffects)
ggplot2::theme_set(theme_bw())
color_1 = '#cc5058'
color_2 = '#6250cc'2. Datos
Los datos corresponden a un caso conocido en términos de modelos de clasificación. La idea es utilizar un dataset simple, con efectos “esperables” de cada variable, para entender mejor los conceptos.
Se busca estimar la probabilidad de que un pasajero del Titanic sobreviva al hundimiento, dadas sus características: edad, género, tarifa, tipo del boleto, entre otras.
Un primer aspecto relevante en este tipo de modelos es la definición de la variable a predecir. En este caso, se busca predecir cuán probable es que un individuo sobreviva al hundimiento. Para ello, se transforma a la variable de supervivencia (survived) en un factor con 2 niveles (0 y 1). El nivel 1 es el segundo nivel. Esto es importante para que los coeficientes estimados en la regresión sean interpretados correctamente: un coeficiente positivo indicaría que determinada variable incrementa la probabilidad de supervivencia.
Al visualizar los niveles de la variable de tipo factor, se observa que el primer nivel es 0 y el segundo es 1.
3. Análisis exploratorio
El entendimiento de los datos es fundamental para ajustar cualquier modelo. En este caso, al ser un modelo de clasificación se realiza un análisis exploratorio general y luego un análisis específico, en relación a la variable a predecir: survived.
📊EDA general
EDA
Seleccionar las distintas opciones para análisis exploratorio general.
{skimr} 📦
{skimr}1 permite realizar un análisis exploratorio global con una función:
Show code
skim(df)| Name | df |
| Number of rows | 891 |
| Number of columns | 12 |
| _______________________ | |
| Column type frequency: | |
| character | 3 |
| factor | 3 |
| numeric | 6 |
| ________________________ | |
| Group variables | None |
Variable type: character
| skim_variable | n_missing | complete_rate | min | max | empty | n_unique | whitespace |
|---|---|---|---|---|---|---|---|
| name | 0 | 1.00 | 12 | 82 | 0 | 891 | 0 |
| ticket | 0 | 1.00 | 3 | 18 | 0 | 681 | 0 |
| cabin | 687 | 0.23 | 1 | 15 | 0 | 147 | 0 |
Variable type: factor
| skim_variable | n_missing | complete_rate | ordered | n_unique | top_counts |
|---|---|---|---|---|---|
| survived | 0 | 1 | FALSE | 2 | 0: 549, 1: 342 |
| sex | 0 | 1 | FALSE | 2 | mal: 577, fem: 314 |
| embarked | 2 | 1 | FALSE | 3 | S: 644, C: 168, Q: 77 |
Variable type: numeric
| skim_variable | n_missing | complete_rate | mean | sd | p0 | p25 | p50 | p75 | p100 | hist |
|---|---|---|---|---|---|---|---|---|---|---|
| passenger_id | 0 | 1.0 | 446.00 | 257.35 | 1.00 | 223.50 | 446.00 | 668.5 | 891.00 | ▇▇▇▇▇ |
| pclass | 0 | 1.0 | 2.31 | 0.84 | 1.00 | 2.00 | 3.00 | 3.0 | 3.00 | ▃▁▃▁▇ |
| age | 177 | 0.8 | 29.70 | 14.53 | 0.42 | 20.12 | 28.00 | 38.0 | 80.00 | ▂▇▅▂▁ |
| sib_sp | 0 | 1.0 | 0.52 | 1.10 | 0.00 | 0.00 | 0.00 | 1.0 | 8.00 | ▇▁▁▁▁ |
| parch | 0 | 1.0 | 0.38 | 0.81 | 0.00 | 0.00 | 0.00 | 0.0 | 6.00 | ▇▁▁▁▁ |
| fare | 0 | 1.0 | 32.20 | 49.69 | 0.00 | 7.91 | 14.45 | 31.0 | 512.33 | ▇▁▁▁▁ |
{modelsummary} 📦
{modelsummary}2 es una alternativa muy similar a {skimr}
Show code
modelsummary::datasummary_skim(df)| Unique (#) | Missing (%) | Mean | SD | Min | Median | Max | ||
|---|---|---|---|---|---|---|---|---|
| passenger_id | 891 | 0 | 446.0 | 257.4 | 1.0 | 446.0 | 891.0 | |
| pclass | 3 | 0 | 2.3 | 0.8 | 1.0 | 3.0 | 3.0 | |
| age | 89 | 20 | 29.7 | 14.5 | 0.4 | 28.0 | 80.0 | |
| sib_sp | 7 | 0 | 0.5 | 1.1 | 0.0 | 0.0 | 8.0 | |
| parch | 7 | 0 | 0.4 | 0.8 | 0.0 | 0.0 | 6.0 | |
| fare | 248 | 0 | 32.2 | 49.7 | 0.0 | 14.5 | 512.3 |
Show code
modelsummary::datasummary_skim(df, type="categorical")| N | % | ||
|---|---|---|---|
| survived | 0 | 549 | 61.6 |
| 1 | 342 | 38.4 | |
| sex | female | 314 | 35.2 |
| male | 577 | 64.8 | |
| embarked | C | 168 | 18.9 |
| Q | 77 | 8.6 | |
| S | 644 | 72.3 |
summary
R base incluye una función para análisis exploratorio general: summary()
passenger_id survived pclass name
Min. : 1.0 0:549 Min. :1.000 Length:891
1st Qu.:223.5 1:342 1st Qu.:2.000 Class :character
Median :446.0 Median :3.000 Mode :character
Mean :446.0 Mean :2.309
3rd Qu.:668.5 3rd Qu.:3.000
Max. :891.0 Max. :3.000
sex age sib_sp parch
female:314 Min. : 0.42 Min. :0.000 Min. :0.0000
male :577 1st Qu.:20.12 1st Qu.:0.000 1st Qu.:0.0000
Median :28.00 Median :0.000 Median :0.0000
Mean :29.70 Mean :0.523 Mean :0.3816
3rd Qu.:38.00 3rd Qu.:1.000 3rd Qu.:0.0000
Max. :80.00 Max. :8.000 Max. :6.0000
NA's :177
ticket fare cabin embarked
Length:891 Min. : 0.00 Length:891 C :168
Class :character 1st Qu.: 7.91 Class :character Q : 77
Mode :character Median : 14.45 Mode :character S :644
Mean : 32.20 NA's: 2
3rd Qu.: 31.00
Max. :512.33
Matriz de correlación
Show code
# Matriz de correlaciones de Pearson
R <- recipe(survived ~ ., data = df) %>%
step_rm(name, ticket, cabin) %>%
step_impute_mode(all_nominal_predictors()) %>%
step_impute_median(all_numeric_predictors()) %>%
step_dummy(all_nominal_predictors(), one_hot = TRUE) %>%
prep() %>% juice() %>%
select(where(is.numeric), -passenger_id) %>%
cor()
R %>% corrplot::corrplot(
method = 'circle', type = "lower", tl.cex = 1, addCoef.col = 'black')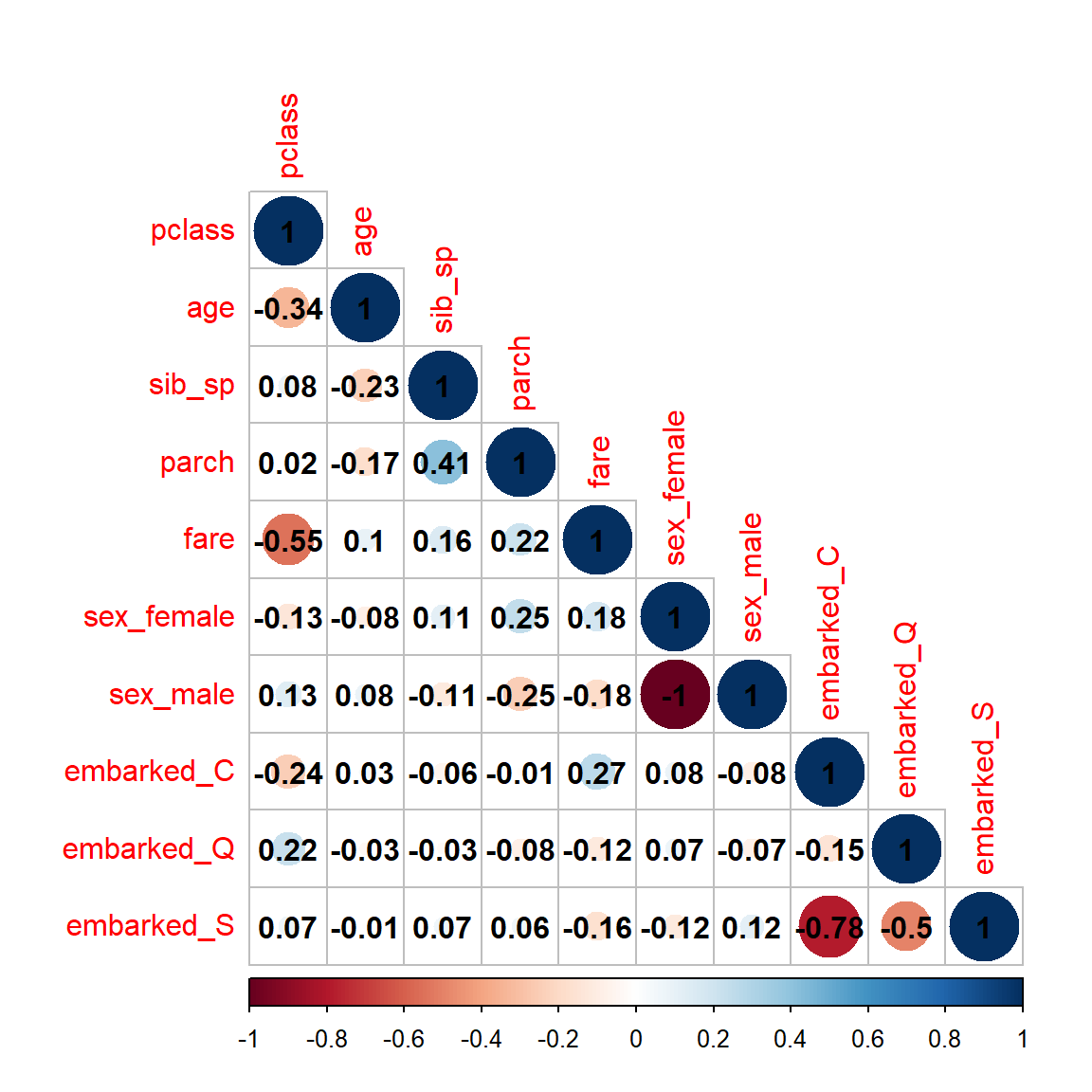
Al incluir todas las categorías en la matriz de correlación, se observa por ejemplo que sex=male y sex=female tiene una correlación = -1, glm detecta esto y en el modelo elimina una de las categorías, ya que es redundante.
🔬EDA para modelos de clasificación
Considerando que se busca predecir la supervivencia, en este caso se presentan algunas alternativas para evaluar la relación de cada variable con la variable a predecir.
EDA
Seleccionar alguna alternativa para visualizar un análisis de cada variable en relación a la variable a predecir (survived).
{gtsummary}📦
{gtsummary}3 define qué estadísticos mostrar en relación al tipo de variable que se considera, aunque también se puede definir manualmente.
Es posible añadir la cantidad de observaciones totales (add_n) y los p-valores (add_p). Los p-valores hacen referencia al tipo de test que se realiza para comparar la variable entre los segmentos analizados. Esto también puede ser definido manualmente.
Show code
tbl_summary(df %>% select(-name, -cabin, -ticket),
by = survived, ) %>%
add_n() %>%
add_p() %>%
modify_header(label = "**Variable**") %>%
bold_labels() | Variable | N | 0, N = 5491 | 1, N = 3421 | p-value2 |
|---|---|---|---|---|
| passenger_id | 891 | 455 (211, 675) | 440 (251, 652) | 0.9 |
| pclass | 891 | <0.001 | ||
| 1 | 80 (15%) | 136 (40%) | ||
| 2 | 97 (18%) | 87 (25%) | ||
| 3 | 372 (68%) | 119 (35%) | ||
| sex | 891 | <0.001 | ||
| female | 81 (15%) | 233 (68%) | ||
| male | 468 (85%) | 109 (32%) | ||
| age | 714 | 28 (21, 39) | 28 (19, 36) | 0.2 |
| Unknown | 125 | 52 | ||
| sib_sp | 891 | |||
| 0 | 398 (72%) | 210 (61%) | ||
| 1 | 97 (18%) | 112 (33%) | ||
| 2 | 15 (2.7%) | 13 (3.8%) | ||
| 3 | 12 (2.2%) | 4 (1.2%) | ||
| 4 | 15 (2.7%) | 3 (0.9%) | ||
| 5 | 5 (0.9%) | 0 (0%) | ||
| 8 | 7 (1.3%) | 0 (0%) | ||
| parch | 891 | <0.001 | ||
| 0 | 445 (81%) | 233 (68%) | ||
| 1 | 53 (9.7%) | 65 (19%) | ||
| 2 | 40 (7.3%) | 40 (12%) | ||
| 3 | 2 (0.4%) | 3 (0.9%) | ||
| 4 | 4 (0.7%) | 0 (0%) | ||
| 5 | 4 (0.7%) | 1 (0.3%) | ||
| 6 | 1 (0.2%) | 0 (0%) | ||
| fare | 891 | 10 (8, 26) | 26 (12, 57) | <0.001 |
| embarked | 889 | <0.001 | ||
| C | 75 (14%) | 93 (27%) | ||
| Q | 47 (8.6%) | 30 (8.8%) | ||
| S | 427 (78%) | 217 (64%) | ||
| Unknown | 0 | 2 | ||
| 1 Median (IQR); n (%) | ||||
| 2 Wilcoxon rank sum test; Pearson’s Chi-squared test; Fisher’s exact test | ||||
{modelsummary} 📦
Con {modelsummary} se puede obtener algo similar:
Show code
modelsummary::datasummary_balance( ~ survived,
data = df)| Mean | Std. Dev. | Mean | Std. Dev. | ||
|---|---|---|---|---|---|
| passenger_id | 447.0 | 260.6 | 444.4 | 252.4 | |
| pclass | 2.5 | 0.7 | 2.0 | 0.9 | |
| age | 30.6 | 14.2 | 28.3 | 15.0 | |
| sib_sp | 0.6 | 1.3 | 0.5 | 0.7 | |
| parch | 0.3 | 0.8 | 0.5 | 0.8 | |
| fare | 22.1 | 31.4 | 48.4 | 66.6 | |
| N | Pct. | N | Pct. | ||
| sex | female | 81 | 14.8 | 233 | 68.1 |
| male | 468 | 85.2 | 109 | 31.9 | |
| embarked | C | 75 | 13.7 | 93 | 27.2 |
| Q | 47 | 8.6 | 30 | 8.8 | |
| S | 427 | 77.8 | 217 | 63.5 |
4. Partición en train y test
Se realiza una partición del dataframe en 2:
Datos para entrenamiento: 85% de los datos
Datos para evaluación: 15% de los datos
La partición se realiza en forma estratificada por la variable a predecir (survived).
Show code
set.seed(42)
splits <- initial_split(data = df,
prop = 0.85,
strata = survived)5. Preprocesamiento
Tal como se observó en el análisis exploratorio, existen valores faltantes. Se genera una receta de preprocesamiento de datos previo al modelado. Para ello, se utiliza el paquete {recipes} 📦, incluido en {tidymodels}4📦.
Decidí no utilizar los pasos de normalización de variables ni generación de variables dummies para contar con el dataset en el formato más similar al original posible para la interpretación final de los resultados.
Se visualizan los datos luego de las transformaciones:
Rows: 756
Columns: 9
$ passenger_id <dbl> 1, 5, 6, 8, 13, 14, 15, 17, 19, 21, 25, 28, 30,…
$ pclass <dbl> 3, 3, 3, 3, 3, 3, 3, 3, 3, 2, 3, 1, 3, 1, 2, 1,…
$ sex <fct> male, male, male, male, male, male, female, mal…
$ age <dbl> 22, 35, 28, 2, 20, 39, 14, 2, 31, 35, 8, 19, 28…
$ sib_sp <dbl> 1, 0, 0, 3, 0, 1, 0, 4, 1, 0, 3, 3, 0, 0, 0, 1,…
$ parch <dbl> 0, 0, 0, 1, 0, 5, 0, 1, 0, 0, 1, 2, 0, 0, 0, 0,…
$ fare <dbl> 7.2500, 8.0500, 8.4583, 21.0750, 8.0500, 31.275…
$ embarked <fct> S, S, Q, S, S, S, S, Q, S, S, S, S, S, C, S, S,…
$ survived <fct> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,…6. Regresión logística univariada
Se parte de una regresión logística univariada. Tomando el enfoque de {tidymodels} 📦, se define que se utilizará un modelo de regresión logística.
model <- parsnip::logistic_reg() %>%
set_mode('classification') %>%
set_engine('glm')El workflow (o pipeline de modelado) incluye la receta de transformaciones de preprocesamiento y el modelo. En este caso, se toma el preprocesador definido anteriormente, sumando un paso adicional que permite seleccionar una única variable (fare) como variable explicativa del modelo, dado que en este caso se busca definir un modelo univariado.
Se ajusta el modelo a los datos de la partición de entrenamiento.
wf_fit <- wf %>% fit(training(splits))Se extrae la regresión logística del pipeline.
reg_log <- wf_fit %>% extract_fit_engine() Revisión del modelo: coeficientes y performance
Se visualiza el modelo ajustado con distintas alternativas. Notar el parametro exponentiate, que permite visualizar los coeficientes en términos del log(OR) o sobre el OR, siendo OR: odd ratio.
{gtsummary} 📦 Log(OR)
Show code
reg_log %>%
tbl_regression(intercept = TRUE, exponentiate = FALSE) %>%
as_gt() %>%
tab_header(title = 'Regresión logística univariada')| Regresión logística univariada | |||
| Characteristic | log(OR)1 | 95% CI1 | p-value |
|---|---|---|---|
| (Intercept) | -1.0 | -1.2, -0.75 | <0.001 |
| fare | 0.02 | 0.01, 0.02 | <0.001 |
| 1 OR = Odds Ratio, CI = Confidence Interval | |||
{gtsummary} 📦 OR
En términos exponenciales:
- OR>1: dy/dx > 0
- OR<1: dy/dx < 0
Show code
reg_log %>%
tbl_regression(intercept = TRUE, exponentiate = TRUE) %>%
as_gt() %>%
tab_header(title = 'Regresión logística univariada')| Regresión logística univariada | |||
| Characteristic | OR1 | 95% CI1 | p-value |
|---|---|---|---|
| (Intercept) | 0.39 | 0.31, 0.47 | <0.001 |
| fare | 1.02 | 1.01, 1.02 | <0.001 |
| 1 OR = Odds Ratio, CI = Confidence Interval | |||
{modelsummary} 📦
Para obtener las estadísticas de bondad de ajuste del modelo, una alternativa es utilizar {modelsummary}. Tal como se verá más adelante, esta alternativa permite visualizar una lista de modelos.
Show code
modelsummary::modelsummary(
list("Regresión logística univariada" = reg_log)
)| Regresión logística univariada | |
|---|---|
| (Intercept) | −0.951 |
| (0.105) | |
| fare | 0.016 |
| (0.003) | |
| Num.Obs. | 756 |
| AIC | 952.2 |
| BIC | 961.4 |
| Log.Lik. | −474.096 |
| RMSE | 0.46 |
{performance} 📦
Se utiliza el paquete {performance} 📦 para evaluar el modelo calibrado. Este es uno de los paquetes contenidos en el ecosistema {easystats}5.
performance::check_model(reg_log)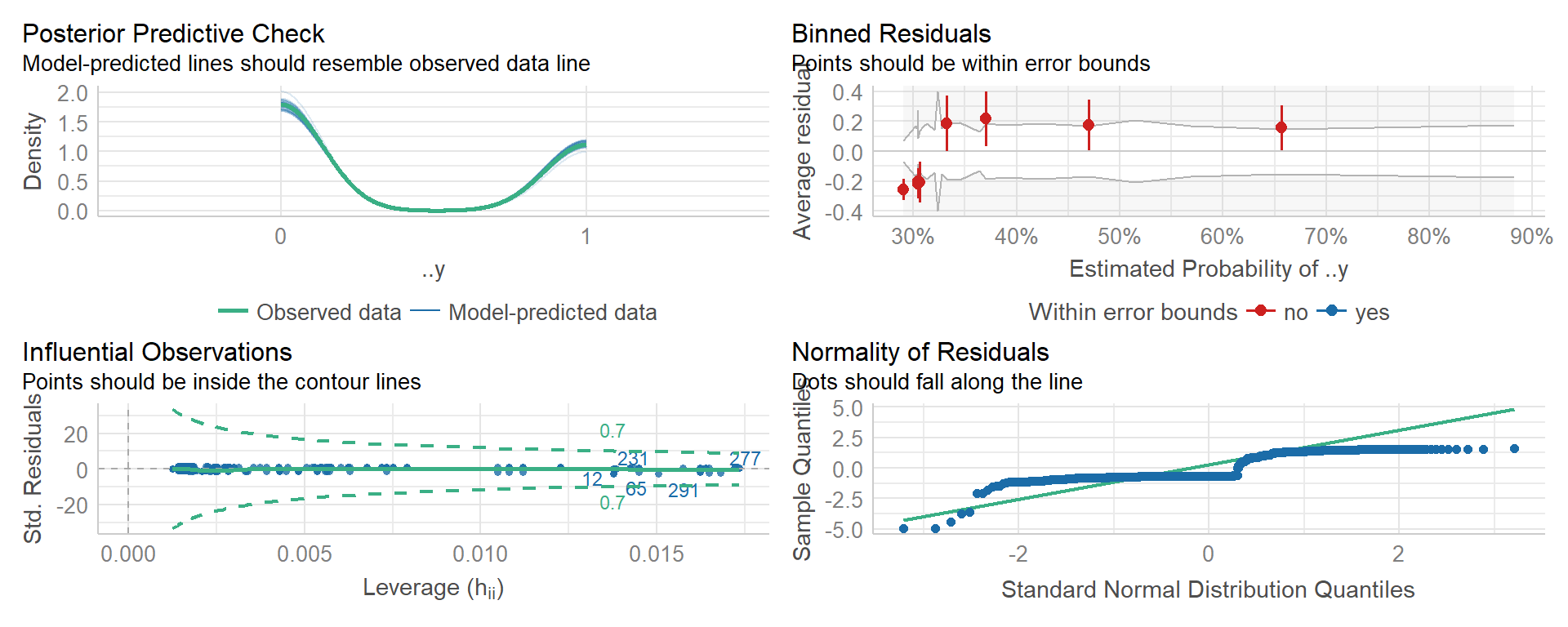
Ecuación
El paquete {equatiomatic}6 📦permite obtener la ecuación del modelo. En este caso, el modelo está definido por:
Show code
reg_log %>%
extract_eq(
var_colors = c(fare = color_2),
greek_colors = c(color_1, color_2),
subscript_colors = c(color_2)
)\[ \log\left[ \frac { P( \operatorname{..y} = \operatorname{1} ) }{ 1 - P( \operatorname{..y} = \operatorname{1} ) } \right] = {\color{#cc5058}{\alpha}} + {\color{#6250cc}{\beta}}_{{\color{#6250cc}{1}}}({\color{#6250cc}{\operatorname{fare}}}) \]
Utilizando use_coef=TRUE, se visualiza la ecuación estimada (con los coeficientes obtenidos del modelo calibrado):
Show code
reg_log %>%
extract_eq(use_coef = TRUE, var_colors = c(fare = color_2))\[ \log\left[ \frac { \widehat{P( \operatorname{..y} = \operatorname{1} )} }{ 1 - \widehat{P( \operatorname{..y} = \operatorname{1} )} } \right] = -0.95 + 0.02({\color{#6250cc}{\operatorname{fare}}}) \]
Inferencias
Dado un modelo ajustado, se realizan inferencias:
predicción de clase: 0=no sobrevivió, 1=sobrevivió
predicción de probabilidad
Show code
| survived | .pred_0 | .pred_1 | .pred_class | fare |
|---|---|---|---|---|
| 1 | 0.70 | 0.30 | 0 | 7.92 |
| 0 | 0.53 | 0.47 | 0 | 51.86 |
| 1 | 0.67 | 0.33 | 0 | 16.70 |
| 0 | 0.70 | 0.30 | 0 | 7.22 |
| 1 | 0.70 | 0.30 | 0 | 7.88 |
| 1 | 0.21 | 0.79 | 1 | 146.52 |
Mediante el coeficiente asociado a la variable fare y el intercepto de la regresión, es posible estimar manualmente la probabilidad de supervivencia. A partir de la ecuación del log(OR) se obtiene la ecuación de P en términos de X:
\[ \log[ \frac {P}{ 1 - P}] = \alpha + \beta_{1}(\operatorname{fare}) \]
\[ \frac {P}{ 1 - P} = e^{\alpha + \beta_{1}(\operatorname{fare})} \]
\[ P = e^{\alpha + \beta_{1}(\operatorname{fare})}(1-P) \]
\[ P = e^{\alpha + \beta_{1}(\operatorname{fare})} - (e^{\alpha + \beta_{1}(\operatorname{fare})})P \]
\[ P + (e^{\alpha + \beta_{1}(\operatorname{fare})})P = e^{\alpha + \beta_{1}(\operatorname{fare})} \]
\[ P[1+(e^{\alpha + \beta_{1}(\operatorname{fare})})] = e^{\alpha + \beta_{1}(\operatorname{fare})} \]
\[ P = \frac{e^{\alpha + \beta_{1}(\operatorname{fare})}}{[1+(e^{\alpha + \beta_{1}(\operatorname{fare})})]} \]
Show code
intercepto <- reg_log$coefficients[['(Intercept)']]
coeficiente <- reg_log$coefficients[['fare']]Notar que en ambos casos se obtienen las mismas predicciones:
Show code
| survived | .pred_1 | .pred_1_manual | fare |
|---|---|---|---|
| 1 | 0.30 | 0.30 | 7.92 |
| 0 | 0.47 | 0.47 | 51.86 |
| 1 | 0.33 | 0.33 | 16.70 |
| 0 | 0.30 | 0.30 | 7.22 |
| 1 | 0.30 | 0.30 | 7.88 |
| 1 | 0.79 | 0.79 | 146.52 |
Visualización de la regresión logística
Para entender el concepto de regresión logística, resulta intuitivo visualizarla gráficamente. En este caso, se presenta en el eje X la variable explicativa (fare) y en el eje Y la variable a predecir (survived). Se muestran 30 observaciones aleatorias con puntos negros. Además, se utiliza stat_smooth() de {ggplot2} 📦para estimar la curva del modelo de regresión logística. Notar que los valores predichos (puntos rojos) caen sobre la curva.
Show code
set.seed(1234)
inferencias %>%
sample_n(10) %>%
ggplot(aes(x = fare, y = as.numeric(survived) - 1)) +
geom_point(alpha = 0.4) +
stat_smooth(
data = training(splits),
method = "glm",
method.args = list(family = "binomial"),
se = FALSE,
aes(color = 'modelo ggplot stat_smooth()')
) +
geom_point(aes(y = .pred_1, color = 'modelo ajustado'),
size = 2,
alpha = 0.6) +
geom_segment(aes(xend = fare, yend = .pred_1),
color = 'black',
linetype = 'dashed') +
scale_color_manual(values = c('blue', 'red')) +
coord_cartesian(xlim = c(0, NA), clip = 'off') +
scale_x_continuous(limits = c(0, NA), expand = c(0, 0)) +
scale_y_continuous(limits = c(0, 1),
expand = c(0, 0),
oob = scales::squish) +
labs(y = 'survived',
title = 'Probabilidad de supervivencia dada la tarifa',
color = 'Modelo') +
theme(plot.margin = unit(c(1, 1, 1, 1), "lines"),
legend.position = 'bottom')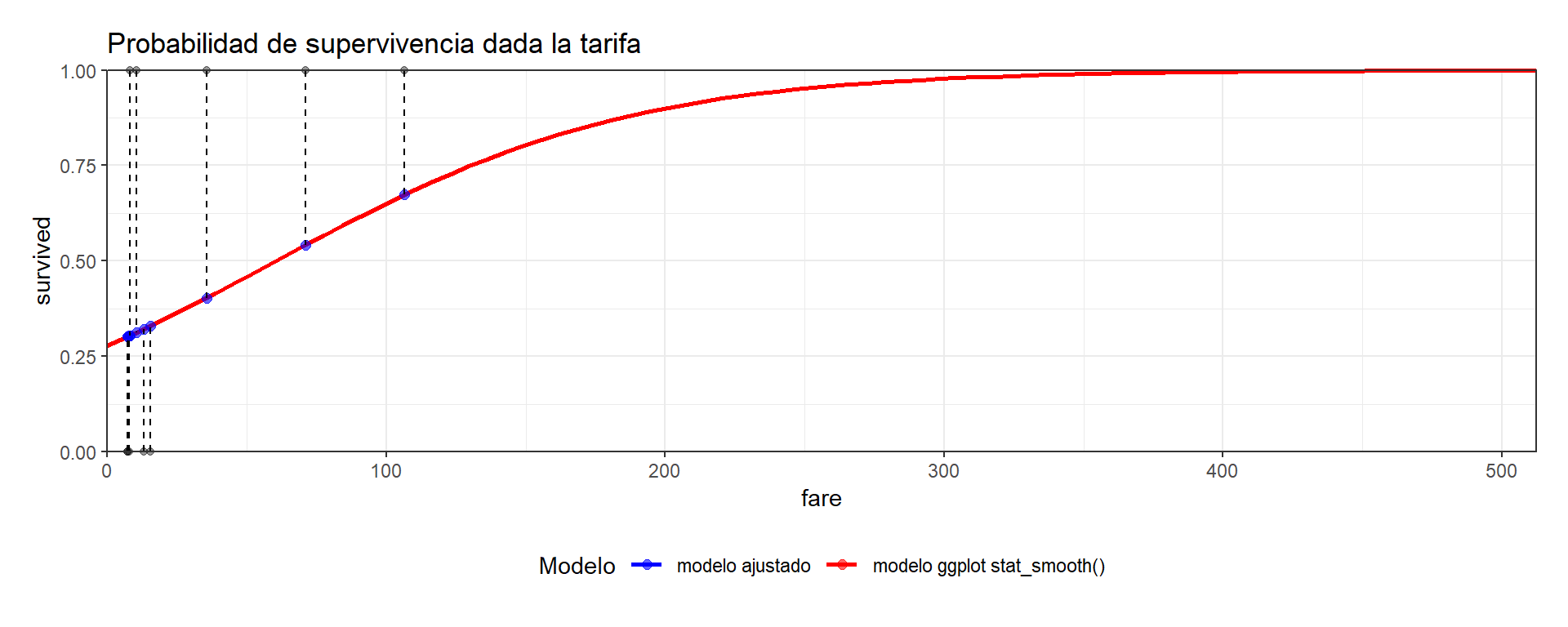
Efectos marginales
Se busca estimar la derivada parcial de la probabilidad estimada ante una variación en X (fare):
\[ dy/dx =\frac{\beta e^{\alpha + \beta{X}}}{[1+e^{(\alpha + \beta{X}))}]^2} \]
El paquete {marginaleffects} 📦 permite estimar este efecto a partir del modelo. Tal como se observó en el gráfico de la regresión logística, la pendiente no es constante a lo largo de la curva. Esto se ve representado en la derivada, que cambia ante distintos valores de X. Por esta razón, para el efecto marginal existen diversas alternativas:
- Evaluar el efecto marginal en un valor de X
- Evaluar el efecto marginal promedio
Efecto marginal dado un valor de X
Se realiza el cálculo manual, asumiendo que la variable X (fare) toma un valor de $200
Show code
valor_x = 100
efecto_marginal <- function(intercepto, coeficiente, valor_x) {
numerador <- coeficiente * exp(intercepto + coeficiente * valor_x)
denominador <- 1 + exp(intercepto + coeficiente * valor_x)
em <- numerador / (denominador * denominador)
return(em)
}
em_100 <- efecto_marginal(intercepto = intercepto,
coeficiente = coeficiente,
valor_x = valor_x)
cat(paste0('Efecto marginal = ',round(em_100,4)))Efecto marginal = 0.0036Se verifica que el resultado obtenido con la función marginaleffects() es equivalente a realizar el cálculo manual de la derivada:
efectos_marginales <- reg_log %>%
marginaleffects(newdata = data.frame(fare = c(valor_x)),
conf_level = 0.95,
slope = 'dydx') %>%
summary()Show code
efectos_marginales %>%
gt() %>%
tab_header(title='Efectos marginales') %>%
fmt_number(where(is.numeric), decimals=4)| Efectos marginales | |||||||
| type | term | estimate | std.error | statistic | p.value | conf.low | conf.high |
|---|---|---|---|---|---|---|---|
| response | fare | 0.0036 | 0.0004 | 9.3035 | 0.0000 | 0.0028 | 0.0043 |
Gráficamente, el efecto marginal en un punto representa la pendiente de la recta tangente a la curva en ese punto. A continuación se visualiza el efecto marginal para 2 valores de la variable fare (50 y 250).
Notar que, si un individuo pagó 50, si hubiera incrementado un poco su pago la probabilidad de supervivencia hubiera aumentado más que si un individuo que pagó 250 hubiera incrementado su pago en la misma cantidad.
Show code
valor_1 = 50
valor_2 = 250
valor_predicho_1 <-
predict(reg_log, data.frame(fare = valor_1), type = 'response')
em_1 <- efecto_marginal(intercepto = intercepto,
coeficiente = coeficiente,
valor_x = valor_1)
valor_predicho_2 <-
predict(reg_log, data.frame(fare = valor_2), type = 'response')
em_2 <- efecto_marginal(intercepto = intercepto,
coeficiente = coeficiente,
valor_x = valor_2)
inferencias %>%
ggplot(aes(x = fare, y = as.numeric(survived) - 1)) +
geom_point(alpha = 0.4) +
stat_smooth(
data = preproc %>% prep() %>% juice(),
method = "glm",
method.args = list(family = "binomial"),
se = FALSE,
show.legend = FALSE,
color = 'red'
) +
geom_vline(xintercept = 0) +
# Caso 1
geom_point(
aes(x = valor_1, y = valor_predicho_1),
color = color_1,
size = 2,
alpha = 0.5
) +
annotate(
"segment",
x = valor_1 ,
y = 0,
xend = valor_1,
yend = valor_predicho_1,
color = color_1,
linetype = 'dashed',
size = 1
) +
annotate(
"segment",
x = 0 ,
y = valor_predicho_1,
xend = valor_1,
yend = valor_predicho_1,
color = color_1,
linetype = 'dashed',
size = 1
) +
geomtextpath::geom_textabline(
label = paste0('dy/dx=', round(em_1, 4)),
color = color_1,
size = 5,
linewidth = 1,
hjust = 0.4,
vjust = -0.2,
intercept = valor_predicho_1 - em_1 * valor_1,
slope = em_1
) +
geom_text(
x = valor_1,
y = valor_predicho_1,
label = paste0('P(survived | x=', valor_1, ')=', round(valor_predicho_1, 2)),
color = color_1,
hjust = -0.1,
vjust = 1,
size = 4
) +
# Caso 2
geom_point(
aes(x = valor_2, y = valor_predicho_2),
color = color_2,
size = 2,
alpha = 0.5
) +
annotate(
"segment",
x = valor_2 ,
y = 0,
xend = valor_2,
yend = valor_predicho_2,
color = color_2,
linetype = 'dashed',
size = 1
) +
annotate(
"segment",
x = 0 ,
y = valor_predicho_2,
xend = valor_2,
yend = valor_predicho_2,
color = color_2,
linetype = 'dashed',
size = 1
) +
geomtextpath::geom_textabline(
label = paste0('dy/dx=', round(em_2, 4)),
color = color_2,
size = 5,
linewidth = 1,
hjust = 0.1,
vjust = -0.2,
intercept = valor_predicho_2 - em_2 * valor_2,
slope = em_2
) +
geom_text(
x = valor_2,
y = valor_predicho_2,
label = paste0('P(survived | x=', valor_2, ')=', round(valor_predicho_2, 2)),
color = color_2,
hjust = -0.1,
vjust = 1,
size = 4
) +
labs(y = 'survived', title = 'P(survived)',
color = 'Modelo') +
theme(plot.margin = unit(c(1, 1, 1, 1), "lines")) +
coord_cartesian(clip = 'on') +
scale_x_continuous(limits = c(0, NA), expand = c(0, 0))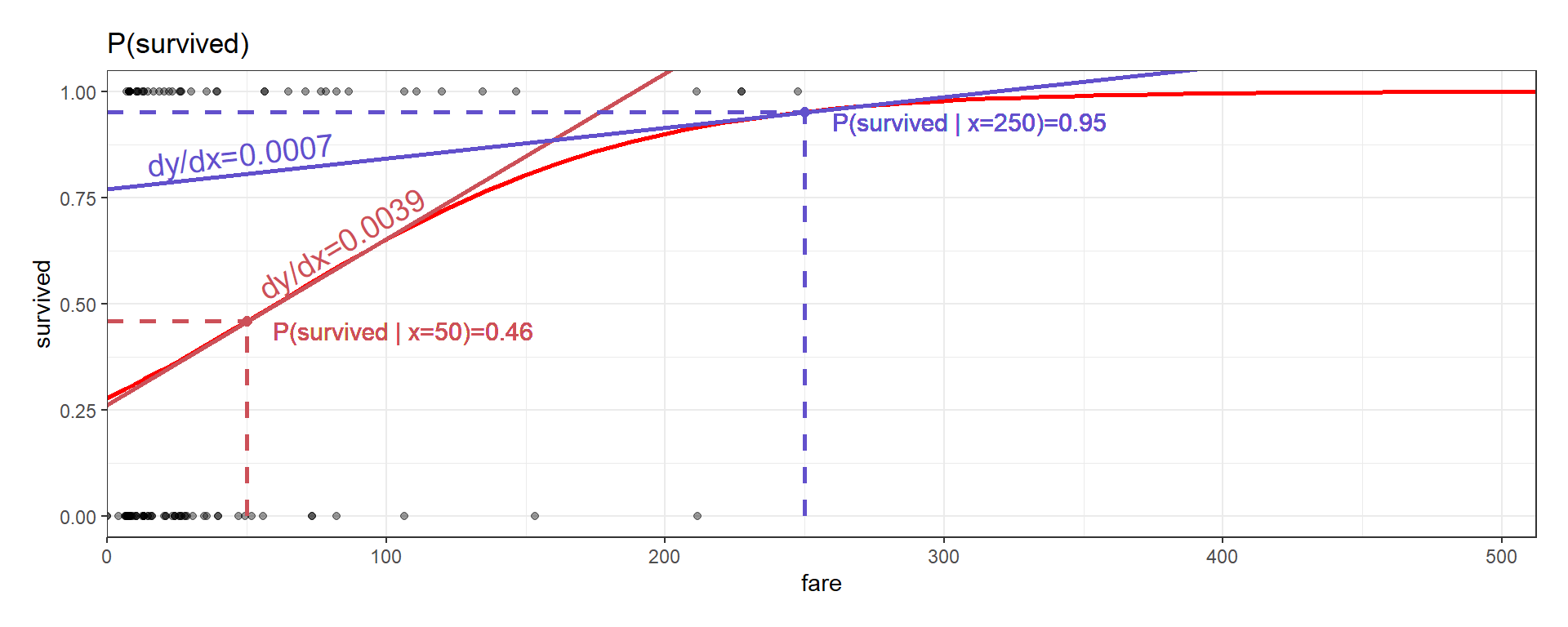
Efecto marginal en la media
Si se utiliza la función para calcular el efecto marginal promedio de la variable fare sobre la probabilidad:
efectos_marginales <- reg_log %>%
marginaleffects(newdata = 'mean',
conf_level = 0.95,
slope = 'dydx') %>% #eyex para elasticidades
summary()Show code
efectos_marginales %>%
gt() %>%
tab_header(title='Efectos marginales') %>%
fmt_number(where(is.numeric), decimals=4)| Efectos marginales | |||||||
| type | term | estimate | std.error | statistic | p.value | conf.low | conf.high |
|---|---|---|---|---|---|---|---|
| response | fare | 0.0037 | 0.0006 | 6.0866 | 0.0000 | 0.0025 | 0.0049 |
Notar que se está considerando el efecto marginal en términos de derivada parcial. Es posible utilizar la función para estimar el efecto marginal en términos de elasticidades o semi-elasticidades (eyex, eydx, dyex).
Existen otros paquetes para calcular efectos marginales, por ejemplo {mfx} 📦. Se observa que los resultados son equivalentes.
Show code
mfx::logitmfx('survived ~ fare', data=df, atmean = TRUE, robust = FALSE)Call:
mfx::logitmfx(formula = "survived ~ fare", data = df, atmean = TRUE,
robust = FALSE)
Marginal Effects:
dF/dx Std. Err. z P>|z|
fare 0.00361165 0.00054088 6.6774 0.00000000002433 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 17.Regresión logística multivariada
Se ajusta el modelo a los datos de la partición de entrenamiento.
wf_fit <- wf %>% fit(training(splits))Se extrae la regresión del pipeline:
reg_log_multivariada <- wf_fit %>% extract_fit_engine()Se visualiza el modelo ajustado. Notar que, en las variables categóricas, una de las categorías es omitida y se estiman los coeficientes restantes.
{modelsummary} 📦
Show code
modelsummary(
list(
"Regresión univariada"=reg_log,
'Regresión multivariada' = reg_log_multivariada
),
estimate = "{estimate} ({std.error}){stars}",
statistic = NULL
)| Regresión univariada | Regresión multivariada | |
|---|---|---|
| (Intercept) | −0.951 (0.105)*** | 5.624 (0.613)*** |
| fare | 0.016 (0.003)*** | 0.001 (0.002) |
| pclass | −1.262 (0.159)*** | |
| sexmale | −2.777 (0.220)*** | |
| age | −0.037 (0.009)*** | |
| sib_sp | −0.282 (0.114)* | |
| parch | −0.084 (0.124) | |
| embarkedQ | 0.052 (0.417) | |
| embarkedS | −0.558 (0.257)* | |
| Num.Obs. | 756 | 756 |
| AIC | 952.2 | 671.5 |
| BIC | 961.4 | 713.1 |
| Log.Lik. | −474.096 | −326.739 |
| RMSE | 0.46 | 0.37 |
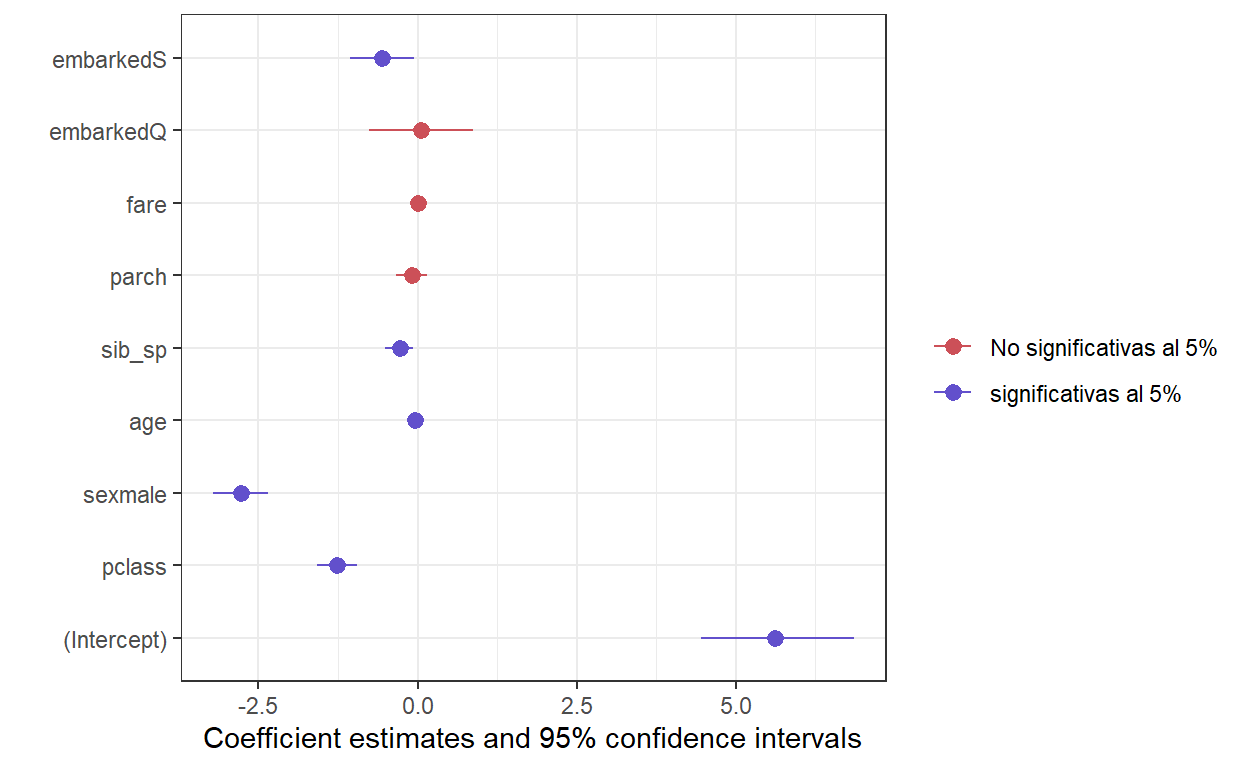
Notar que en el caso multivariado la variable fare ya no es significativa, mientras que en el caso univariado sí lo era. Tal como se observó en la matriz de correlación al inicio, la correlación de la variable fare con la pclass era elevada.
{modelsummary}📦 modelplot
Ecuación
Show code
reg_log_multivariada %>%
extract_eq(use_coef=TRUE, wrap = TRUE, terms_per_line=1)\[ \begin{aligned} \log\left[ \frac { \widehat{P( \operatorname{..y} = \operatorname{1} )} }{ 1 - \widehat{P( \operatorname{..y} = \operatorname{1} )} } \right] &= 5.62\ - \\ &\quad 1.26(\operatorname{pclass})\ - \\ &\quad 2.78(\operatorname{sex}_{\operatorname{male}})\ - \\ &\quad 0.04(\operatorname{age})\ - \\ &\quad 0.28(\operatorname{sib\_sp})\ - \\ &\quad 0.08(\operatorname{parch})\ + \\ &\quad 0(\operatorname{fare})\ + \\ &\quad 0.05(\operatorname{embarked}_{\operatorname{Q}})\ - \\ &\quad 0.56(\operatorname{embarked}_{\operatorname{S}}) \end{aligned} \]
Predicción en la media
apm <- predictions(reg_log_multivariada, newdata = "mean")Show code
apm %>%
gt() %>%
tab_header(title='Predicción en la media') %>%
fmt_number(where(is.numeric), decimals=2)| Predicción en la media | |||||||||||||||
| rowid | type | predicted | std.error | statistic | p.value | conf.low | conf.high | ..y | pclass | sex | age | sib_sp | parch | fare | embarked |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1.00 | response | 0.14 | 0.02 | 7.48 | 0.00 | 0.10 | 0.17 | 0 | 2.31 | male | 29.26 | 0.54 | 0.40 | 31.93 | S |
Efectos marginales
Se busca estimar la derivada parcial de la variable a predecir en relación a cada una de las variables del modelo.
Efecto marginal en la media
efectos_marginales <- reg_log_multivariada %>%
marginaleffects(newdata = 'mean',
conf_level = 0.95,
slope = 'dydx') %>%
summary()Show code
efectos_marginales %>%
gt() %>%
tab_header(title = 'Efectos marginales') %>%
fmt_number(where(is.numeric), decimals = 2)| Efectos marginales | ||||||||
| type | term | contrast | estimate | std.error | statistic | p.value | conf.low | conf.high |
|---|---|---|---|---|---|---|---|---|
| response | pclass | dY/dX | −0.15 | 0.02 | −7.10 | 0.00 | −0.19 | −0.11 |
| response | sex | male - female | −0.58 | 0.04 | −15.37 | 0.00 | −0.65 | −0.51 |
| response | age | dY/dX | 0.00 | 0.00 | −4.02 | 0.00 | −0.01 | 0.00 |
| response | sib_sp | dY/dX | −0.03 | 0.01 | −2.43 | 0.02 | −0.06 | −0.01 |
| response | parch | dY/dX | −0.01 | 0.01 | −0.68 | 0.50 | −0.04 | 0.02 |
| response | fare | dY/dX | 0.00 | 0.00 | 0.49 | 0.63 | 0.00 | 0.00 |
| response | embarked | Q - C | 0.01 | 0.07 | 0.12 | 0.90 | −0.13 | 0.15 |
| response | embarked | S - C | −0.08 | 0.04 | −1.94 | 0.05 | −0.16 | 0.00 |
Notar que, en este caso, se observan efectos marginales para variables numéricas y categóricas:
El efecto marginal de las variables numéricas, en el caso multivariado, es equivalente al mencionado en el caso univariado.
En las variables categóricas se analiza la diferencia en probabilidad ante un cambio de categoría.
El efecto marginal en la media es equivalente a tomar la observación promedio (apm, calculada anteriormente) y verificar el efecto marginal en ese punto (efecto marginal en la media):
Efecto marginal promedio
El promedio del efecto marginal de cada variable para cada observación del df:
efectos_marginales <- reg_log_multivariada %>%
marginaleffects(newdata = training(splits),
conf_level = 0.95,
slope = 'dydx') %>%
summary()Show code
efectos_marginales %>%
gt() %>%
tab_header(title = 'Efectos marginales') %>%
fmt_number(where(is.numeric), decimals = 2)| Efectos marginales | ||||||||
| type | term | contrast | estimate | std.error | statistic | p.value | conf.low | conf.high |
|---|---|---|---|---|---|---|---|---|
| response | pclass | dY/dX | −0.17 | 0.02 | −9.02 | 0.00 | −0.21 | −0.14 |
| response | sex | male - female | −0.50 | 0.03 | −15.00 | 0.00 | −0.56 | −0.43 |
| response | age | dY/dX | −0.01 | 0.00 | −4.50 | 0.00 | −0.01 | 0.00 |
| response | sib_sp | dY/dX | −0.04 | 0.02 | −2.51 | 0.01 | −0.07 | −0.01 |
| response | parch | dY/dX | −0.01 | 0.02 | −0.68 | 0.50 | −0.04 | 0.02 |
| response | fare | dY/dX | 0.00 | 0.00 | 0.49 | 0.62 | 0.00 | 0.00 |
| response | embarked | Q - C | 0.01 | 0.06 | 0.12 | 0.90 | −0.11 | 0.13 |
| response | embarked | S - C | −0.08 | 0.04 | −2.13 | 0.03 | −0.15 | −0.01 |
Efecto marginal en un Punto
En este caso, se estiman los efectos marginales para una observación aleatoria.
| passenger_id | survived | pclass | name | sex | age | sib_sp | parch | ticket | fare | cabin | embarked |
|---|---|---|---|---|---|---|---|---|---|---|---|
| 306 | 1 | 1 | Allison, Master. Hudson Trevor | male | 0.92 | 1 | 2 | 113781 | 151.55 | C22 C26 | S |
efectos_marginales <- reg_log_multivariada %>%
marginaleffects(newdata = sample_data,
conf_level = 0.95,
slope = 'dydx') %>%
summary()Show code
efectos_marginales %>%
gt() %>%
tab_header(title = 'Efectos marginales en un punto') %>%
fmt_number(where(is.numeric), decimals = 2)| Efectos marginales en un punto | ||||||||
| type | term | contrast | estimate | std.error | statistic | p.value | conf.low | conf.high |
|---|---|---|---|---|---|---|---|---|
| response | pclass | dY/dX | −0.28 | 0.05 | −6.06 | 0.00 | −0.37 | −0.19 |
| response | sex | male - female | −0.30 | 0.09 | −3.46 | 0.00 | −0.46 | −0.13 |
| response | age | dY/dX | −0.01 | 0.00 | −5.68 | 0.00 | −0.01 | −0.01 |
| response | sib_sp | dY/dX | −0.06 | 0.03 | −2.45 | 0.01 | −0.11 | −0.01 |
| response | parch | dY/dX | −0.02 | 0.03 | −0.65 | 0.52 | −0.07 | 0.04 |
| response | fare | dY/dX | 0.00 | 0.00 | 0.51 | 0.61 | 0.00 | 0.00 |
| response | embarked | Q - C | 0.01 | 0.07 | 0.13 | 0.90 | −0.13 | 0.14 |
| response | embarked | S - C | −0.11 | 0.05 | −2.02 | 0.04 | −0.22 | 0.00 |
Se busca estimar manualmente el efecto marginal de las variables categóricas. Para ello, se utiliza el modelo para estimar la probabilidad en 3 casos:
Observación con valores originales
Observación modificando la categoría sex por “female”, manteniendo el resto de las variables constantes.
Observación modificando la categoría embarked por “C”, manteniendo el resto de las variables constantes.
Show code
p_original <- predict(
reg_log_multivariada, sample_data,
type='response'
)
p_sex <- predict(
reg_log_multivariada, sample_data %>% mutate(sex='female'),
type='response'
)
p_embarked <- predict(
reg_log_multivariada, sample_data %>% mutate(embarked='C'),
type='response'
)
cat(
'P(survived | sex=male & embarked=S)=', round(p_original,2),
'\nP(survived | sex=female)=', round(p_sex,2),
'\nP(survived | embarked=c)=', round(p_embarked,2)
)P(survived | sex=male & embarked=S)= 0.67
P(survived | sex=female)= 0.97
P(survived | embarked=c)= 0.78Por diferencia, se obtienen los efectos marginales, equivalentes a los estimados con {marginaleffects}.
Predicciones condicionales ajustadas
{marginaleffects} 📦 también incluye una función para visualizar las predicciones ajustadas en relación a uno o más predictores.
1 variable
2 variables
Show code
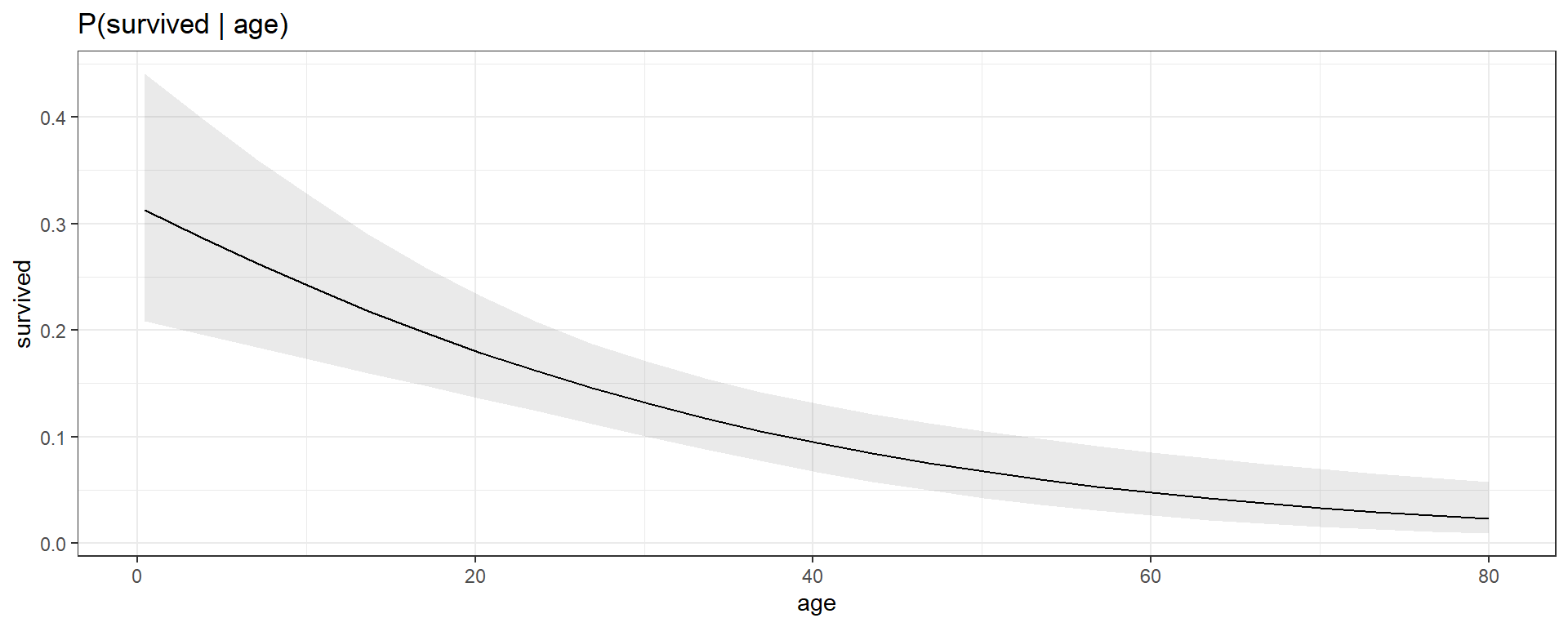
plot_cap(reg_log_multivariada, condition = c("age", "sex"),
type='response', conf_level=0.95)+
scale_color_manual(values=c(color_1, color_2))+
labs(title='P(survived | age, sex)',y='survived')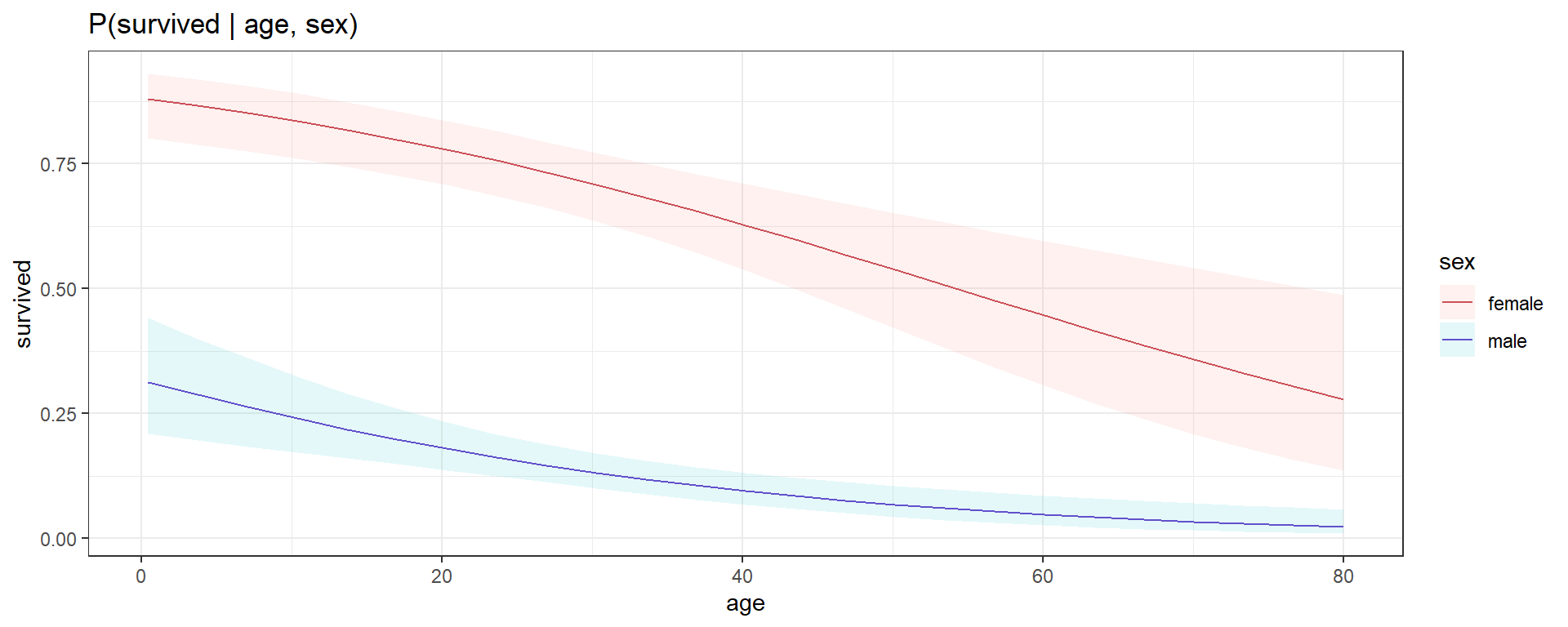
3 variables
Show code
plot_cap(reg_log_multivariada, condition = c("age", "sex", "pclass"),
type='response', conf_level=0.95)+
scale_color_manual(values=c(color_1, color_2))+
labs(title='P(survived | age, sex, pclass)',y='survived')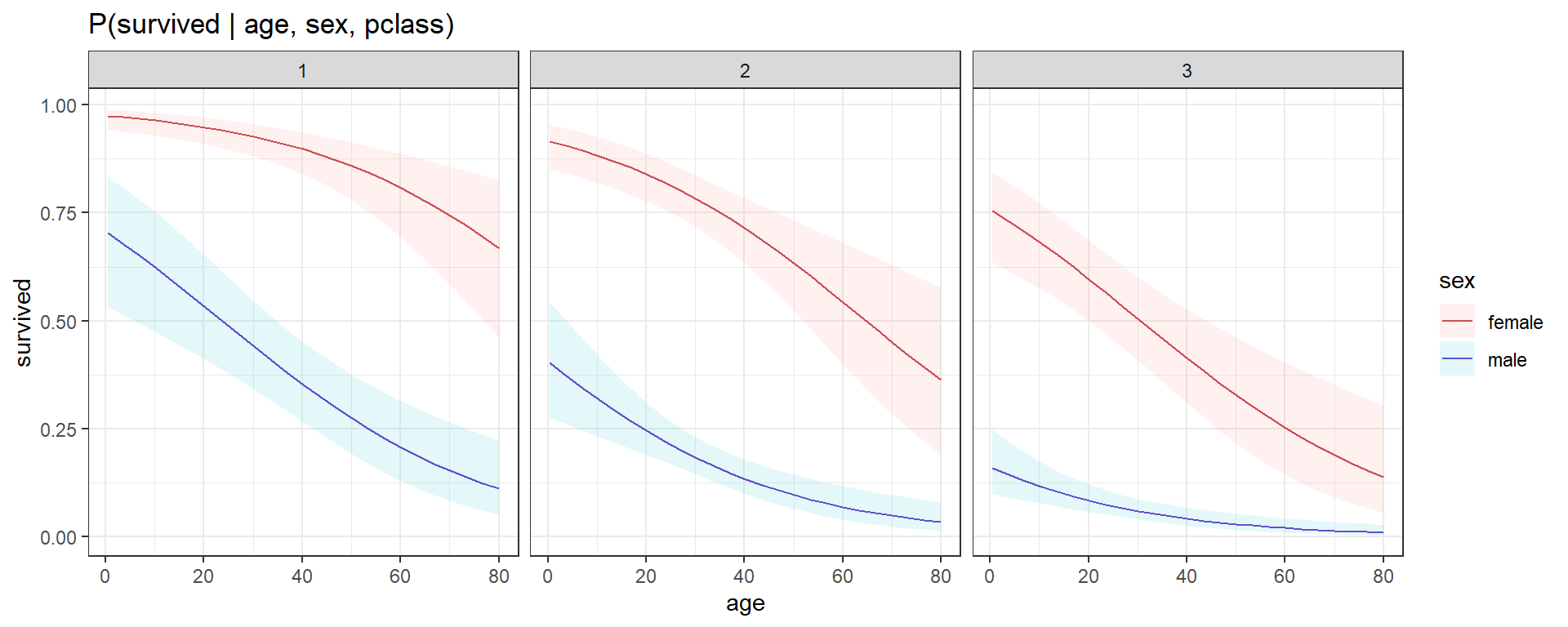
8. Estandarización de datos
Si se busca entender cuáles son las variables que más impactan sobre la supervivencia, es necesario aplicar una estandarización de los datos.. Para realizar la estandarización, se utiliza la función step_normalize() de {recipes} que aplica la siguiente transformación sobre cada una de las variables explicativas:
\[ Z = \frac{X - \mu}{\sigma} \]
Siendo μ = promedio, σ = desvío estándar.
Show code
modelsummary(
list(
"Regresión univariada" = reg_log,
'Regresión multivariada' = reg_log_multivariada,
"Regresión multivariada con normalización" = reg_log_norm
),
estimate = "{estimate} ({std.error}){stars}",
statistic = NULL
)| Regresión univariada | Regresión multivariada | Regresión multivariada con normalización | |
|---|---|---|---|
| (Intercept) | −0.951 (0.105)*** | 5.624 (0.613)*** | 1.482 (0.265)*** |
| fare | 0.016 (0.003)*** | 0.001 (0.002) | 0.061 (0.123) |
| pclass | −1.262 (0.159)*** | −1.053 (0.132)*** | |
| sexmale | −2.777 (0.220)*** | −2.777 (0.220)*** | |
| age | −0.037 (0.009)*** | −0.477 (0.111)*** | |
| sib_sp | −0.282 (0.114)* | −0.322 (0.130)* | |
| parch | −0.084 (0.124) | −0.070 (0.103) | |
| embarkedQ | 0.052 (0.417) | 0.052 (0.417) | |
| embarkedS | −0.558 (0.257)* | −0.558 (0.257)* | |
| Num.Obs. | 756 | 756 | 756 |
| AIC | 952.2 | 671.5 | 671.5 |
| BIC | 961.4 | 713.1 | 713.1 |
| Log.Lik. | −474.096 | −326.739 | −326.739 |
| RMSE | 0.46 | 0.37 | 0.37 |
Notar que:
Los coeficientes asociados a variables categóricas no cambiaron, dado que estas variables se mantienen en su formato original: entran al modelo como dummies (0,1).
Los coeficientes de las variables numéricas tienen ciertos cambios en magnitud, ya que ahora representan la variación en log(OD) en relación al incremento en una únidad en la variable numérica normalizada.
AIC, BIC, Log-likelihood, RMSE, se mantienen todos iguales.
Comentarios finales
En este post mostraron algunas cuestiones vinculadas a regresiones logísticas en R. Cualquier comentario, duda o sugerencia es bienvenida!
Contacto ✉
Karina Bartolome, Linkedin, Twitter, Github, Blogpost
SessionInfo()
Show code
sessioninfo::package_info() %>%
filter(attached==TRUE) %>%
select(package, loadedversion, date, source) %>%
gt() %>%
tab_header(title='Paquetes utilizados',
subtitle='Versiones') %>%
opt_align_table_header('left')| Paquetes utilizados | |||
| Versiones | |||
| package | loadedversion | date | source |
|---|---|---|---|
| broom | 1.0.4 | 2023-03-11 | CRAN (R 4.2.3) |
| dials | 0.1.1 | 2022-04-06 | CRAN (R 4.2.0) |
| dplyr | 1.1.1 | 2023-03-22 | CRAN (R 4.2.3) |
| equatiomatic | 0.3.1 | 2022-01-30 | CRAN (R 4.2.2) |
| forcats | 1.0.0 | 2023-01-29 | CRAN (R 4.2.3) |
| ggplot2 | 3.4.2 | 2023-04-03 | CRAN (R 4.2.0) |
| gt | 0.9.0 | 2023-03-31 | CRAN (R 4.2.3) |
| gtsummary | 1.6.1 | 2022-06-22 | CRAN (R 4.2.1) |
| infer | 1.0.0 | 2021-08-13 | CRAN (R 4.2.0) |
| lubridate | 1.9.2 | 2023-02-10 | CRAN (R 4.2.3) |
| marginaleffects | 0.8.1 | 2022-11-23 | CRAN (R 4.2.2) |
| modeldata | 0.1.1 | 2021-07-14 | CRAN (R 4.2.0) |
| modelsummary | 1.3.0 | 2023-01-05 | CRAN (R 4.2.2) |
| parsnip | 1.0.3 | 2022-11-11 | CRAN (R 4.2.2) |
| purrr | 1.0.1 | 2023-01-10 | CRAN (R 4.2.3) |
| readr | 2.1.4 | 2023-02-10 | CRAN (R 4.2.3) |
| recipes | 1.0.3 | 2022-11-09 | CRAN (R 4.2.2) |
| rsample | 0.1.1 | 2021-11-08 | CRAN (R 4.2.0) |
| scales | 1.2.1 | 2022-08-20 | CRAN (R 4.2.3) |
| skimr | 2.1.4 | 2022-04-15 | CRAN (R 4.2.0) |
| stringr | 1.5.0 | 2022-12-02 | CRAN (R 4.2.3) |
| tibble | 3.2.1 | 2023-03-20 | CRAN (R 4.2.3) |
| tidymodels | 0.2.0 | 2022-03-19 | CRAN (R 4.2.0) |
| tidyr | 1.3.0 | 2023-01-24 | CRAN (R 4.2.3) |
| tidyverse | 2.0.0 | 2023-02-22 | CRAN (R 4.2.3) |
| tune | 0.2.0 | 2022-03-19 | CRAN (R 4.2.0) |
| workflows | 0.2.6 | 2022-03-18 | CRAN (R 4.2.0) |
| workflowsets | 0.2.1 | 2022-03-15 | CRAN (R 4.2.0) |
| yardstick | 1.0.0 | 2022-06-06 | CRAN (R 4.2.0) |
Show code
knitr::knit_exit()